
Кредит: Природные протоколы (2024). DOI: 10.1038/s41596-024-01046-3
Исследовательская группа под руководством ученых из Калифорнийского университета в Риверсайде разработала вычислительный процесс для анализа больших наборов данных в области метаболомики, изучения малых молекул, обнаруженных в клетках, биожидкостях, тканях и целых экосистемах.
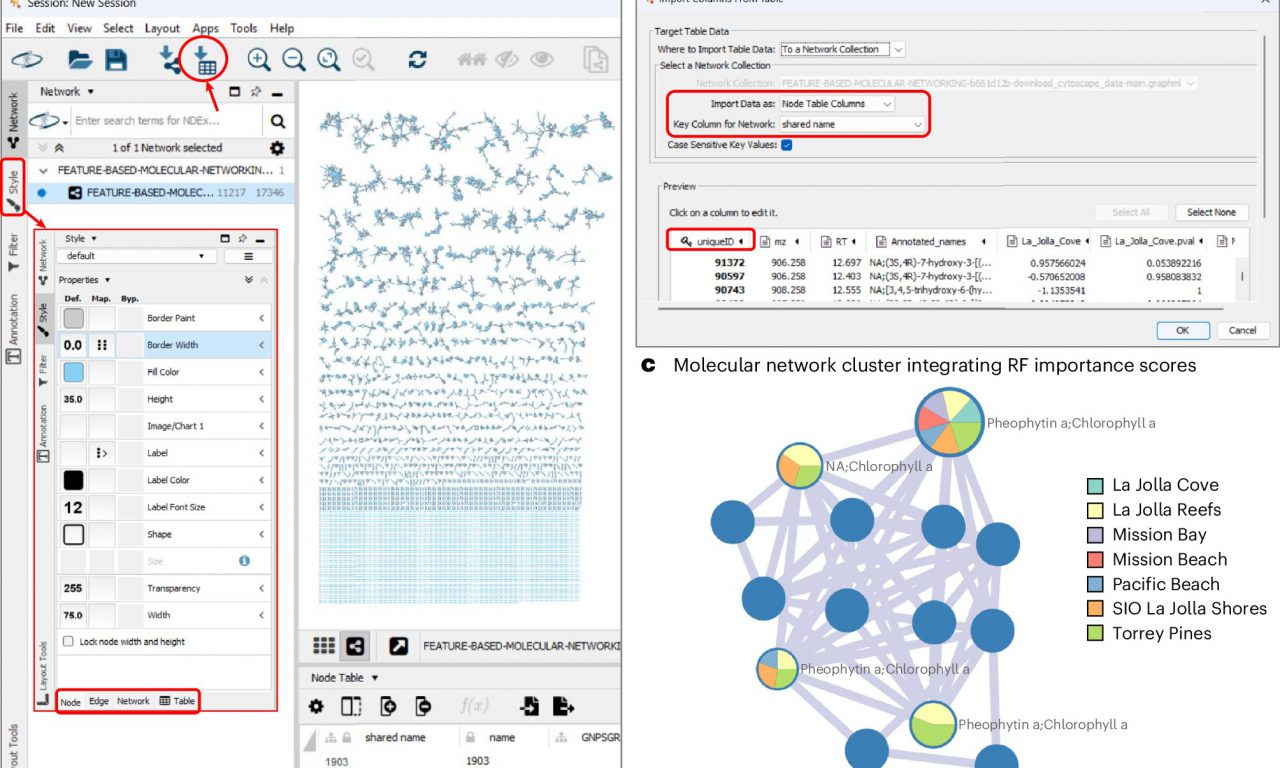
Совсем недавно команда применила этот новый вычислительный инструмент для анализа загрязнителей в морской воде в Южной Калифорнии. Команда быстро составила химический профиль прибрежной среды и выявила потенциальные источники загрязнения.
«Мы заинтересованы в понимании того, как такие загрязнители попадают в экосистему», — сказал Дэниел Петрас, доцент кафедры биохимии Калифорнийского университета в Риверсайде, возглавлявший исследовательскую группу. «Определить, какие молекулы в океане важны для здоровья окружающей среды, непросто из-за огромного химического разнообразия океана. Разработанный нами протокол значительно ускоряет этот процесс. Более эффективная сортировка данных означает, что мы можем быстрее понять проблемы, связанные с загрязнением океана. .»
Пятрас и его коллеги сообщают в журнале Природные протоколы что их протокол предназначен не только для опытных исследователей, но и для образовательных целей, что делает его идеальным ресурсом для студентов и начинающих ученых. Этот вычислительный рабочий процесс сопровождается доступным веб-приложением с графическим пользовательским интерфейсом, которое делает анализ метаболомических данных доступным для неспециалистов и позволяет им получить статистическую информацию о своих данных в течение нескольких минут.
«Этот инструмент доступен широкому кругу исследователей, от абсолютных новичков до экспертов, и предназначен для использования в сочетании с программным обеспечением для молекулярных сетей, которое разрабатывает моя группа», — сказал соавтор Минсюнь Ван, доцент кафедры компьютерных наук и инженерное дело в УЦР. «Для новичков рекомендации и код, которые мы предоставляем, облегчают понимание общих этапов обработки и анализа данных. Для экспертов это ускоряет воспроизводимый анализ данных, позволяя им делиться своими рабочими процессами и результатами статистического анализа данных».
Петрас объяснил, что исследовательская работа уникальна и представляет собой большой образовательный ресурс, организованный виртуальной исследовательской группой под названием Virtual Multiomics Lab (VMOL). VMOL представляет собой сообщество с открытым доступом, в котором участвуют более 50 ученых со всего мира. Его цель – упростить и демократизировать процесс химического анализа, сделав его доступным для исследователей во всем мире, независимо от их опыта и ресурсов.
«Я невероятно горжусь тем, что этот проект превратился во что-то впечатляющее, в нем приняли участие эксперты и студенты со всего мира», — сказал Абзер Паккир Шах, аспирант из группы Петраса и первый автор статьи. «Устраняя физические и экономические барьеры, VMOL обеспечивает обучение вычислительной масс-спектрометрии и науке о данных и стремится запустить виртуальные исследовательские проекты как новую форму совместной науки».
Все программное обеспечение, разработанное командой, бесплатно и общедоступно. Разработка программного обеспечения была начата во время летней школы по нецелевой метаболомике в 2022 году в Тюбингенском университете, где команда также запустила VMOL.
Петрас ожидает, что протокол будет особенно полезен исследователям окружающей среды, а также ученым, работающим в области биомедицины, и исследователям, проводящим клинические исследования в области микробиома.
«Универсальность нашего протокола распространяется на широкий спектр областей и типов образцов, включая комбинаторную химию, анализ допинга и следовые загрязнения пищевых продуктов, фармацевтических препаратов и других промышленных продуктов», — сказал он.
Пятрас получил степень магистра биотехнологии в Университете прикладных наук Дармштадта и докторскую степень по биохимии в Техническом университете Берлина. Он проводил постдокторские исследования в Калифорнийском университете в Сан-Диего, где сосредоточился на разработке крупномасштабных методов метаболомики окружающей среды. В 2021 году он открыл лабораторию функциональной метаболомики в Тюбингенском университете. В январе 2024 года он присоединился к UCR, где его лаборатория занимается разработкой и применением методов масс-спектрометрии для визуализации и оценки химического обмена внутри микробных сообществ.
Дополнительная информация:
Абзер К. Паккир Шах и др., Статистический анализ результатов молекулярных сетей на основе признаков на основе нецелевых данных метаболомики, Природные протоколы (2024). DOI: 10.1038/s41596-024-01046-3
Предоставлено Калифорнийским университетом в Риверсайде.
Цитирование : Новый инструмент обработки данных значительно ускоряет молекулярный анализ нашей окружающей среды (20 сентября 2024 г.), получено 20 сентября 2024 г. с https://phys.org/news/2024-09-science-tool-greatly-molecular-anaлиз.html.
Этот документ защищен авторским правом. За исключением любых добросовестных сделок в целях частного изучения или исследования, никакая часть не может быть воспроизведена без письменного разрешения. Содержимое предоставлено исключительно в информационных целях.
 EUROPEAN UNION
EUROPEAN UNION 